
|
高準確度的長讀取基因定序技術「HiFi 定序」帶來哪些突破與進展? |
|||
|
如同華盛頓大學醫學院 Evan E Eichler 教授在其最近刊載於《Nature Reviews Genetics》期刊的文章中所說的,長讀取定序 (Long-read sequencing) 技術的出現,讓我們得以用新的角度與深度來剖析了解生物體內的 DNA 與 RNA 序列 [1]。以下我們就帶您快速了解長讀取基因定序技術帶來哪些突破與進展?
|
|||
|
相較於 NGS (Next-generation sequencing) 次世代定序技術每一個定序片段長度僅有 <300 bp、且需要仰賴 PCR 擴增技術放大 DNA 樣本,長讀取定序技術每一個定序片段長度可長達 10 kb 至數 Mb,且樣本不需經過 PCR 擴增放大。長讀取定序技術的出現,讓我們首次得以探尋包含端粒 (Telomeres) 等重複序列區域(另有著絲粒 (Centromeres) 與高 GC 含量區域等),並藉由更精確的單倍體 (Haplotype) 全長序列資訊,發掘出許多過去未曾能被發現的序列變異,特別是基因體結構變異 (Structural variants, SVs) [1-4]。
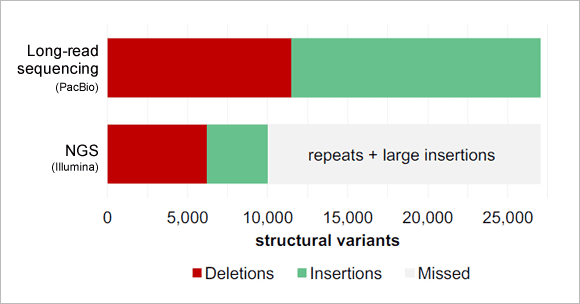
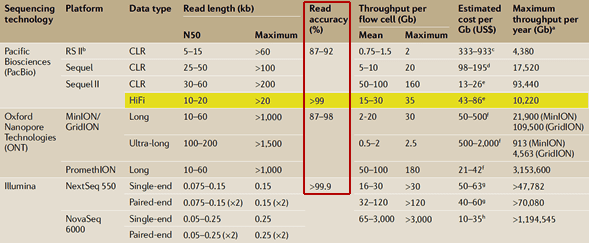
圖 1﹑相較於 NGS 技術,長讀取定序技術可檢測出更多的 SVs。根據 Chaisson 研究團隊的實驗結果,長讀取定序技術檢測出的 SVs 數量約為 NGS 技術的近 2.5 倍 [4]。IMAGE © Pacific Biosciences. 根據 Chaisson 研究團隊的評比實驗結果,長讀取定序技術檢測出的 SVs 數量約為 NGS 技術的近 2.5 倍 (圖 1),絕大部分的 SVs 無法被 NGS 所測得 [4]。這是因為 SVs 這類基因體序列變異具有較長的序列影響長度(≥50 bp),且變異發生位置往往座落在重複序列範圍內,這些特性都導致 SVs 不易被短讀序 (Short read) NGS 技術所偵測得到 [1-4]。而隨著長讀取定序技術的躍進,已有越來越多的人類罕見疾病相關 SVs 相繼被披露,包含神經發展障礙症 (Neurodevelopmental disorders, NDDS)、性聯遺傳肌張力障礙帕金森症候群 (X-linked dystonia-parkinsonism, XDP)、肌萎縮性側索硬化症 (Amyotrophic lateral sclerosis, ALS)、肌抽躍型發作癲癇 (Myoclonic epilepsy)、肌肉強直症 (Myotonic dystrophy)、X 染色體脆折症 (Fragile X syndrome) 等 [1, 2, 5]。 長讀取定序技術在引領我們更全面精準地掌握基因體序列 (Whole-genome sequence) 資訊的同時 [6. 7],也加速推動了我們對轉錄體 (Transcriptome)、RNA 異構物 (RNA isoforms) [8, 9, 10]、複雜基因區域(例如 HLA 基因)[11]、以及表觀遺傳修飾 [12, 13] 有更深入精確的了解。 當前定序技術最新評比結果 目前最具代表性的長讀取定序技術分別來自 Pacific Biosciences (PacBio) 與 Oxford Nanopore Technologies (ONT) 公司。華盛頓大學醫學院 Evan E Eichler 教授研究團隊比較了這兩種長讀取定序技術與 NGS 在定序準確度 (Accuracy)、通量 (Throughput) 與成本花費 (Cost) 的差異。從表格中,我們可以看到 NGS 作為目前主流的定序技術,具有高達 >99.9% 的準確度;而與其準確度最為接近的長讀取定序技術則是來自 PacBio 公司的 HiFi (High fidelity) 定序結果,其準確度為 >99% [1]。
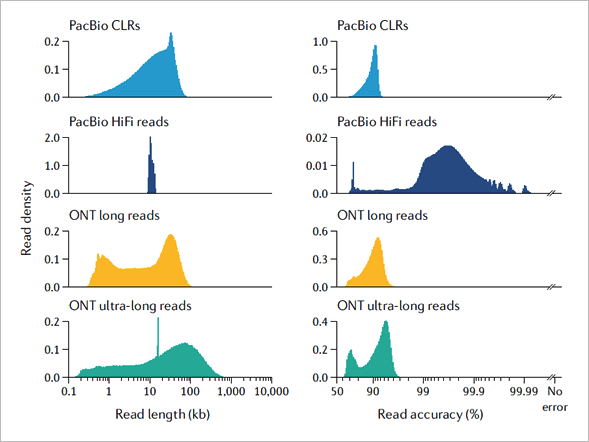
表 1﹑PacBio 公司的 HiFi 定序結果準確度高達 >99%,是最接近 NGS 準確度的長讀取定序技術 [1]。IMAGE © Nat Rev Genet. 2020 Oct;21(10):597-614. Table 1. 作者們進一步比較 PacBio 與 ONT 各個定序方式在定序長度與準確度的差異性。從圖 2 中可以看出,儘管 ONT 具有較長的定序長度,然而其準確度卻無法得到有效提升,離 Q20(即 99%)仍有一段相當大的距離。作者們在文中即表示 "PacBio HiFi sequence reads... are the first data type that is both long (greater than 10 kb in length) and highly accurate (greater than 99%)." [1]。
圖 2﹑儘管 ONT 具有較長的定序長度,然而 PacBio 公司 HiFi 定序結果卻是唯一可以兼顧長讀值與高準確度 (>99%) 的長讀取定序技術 [1]。IMAGE © Nat Rev Genet. 2020 Oct;21(10):597-614. Fig. 3c. HiFi 長讀取定序技術 — 強大便利的研究工具
本篇內容源自 PacBio 與 Gene Company 公司,伯森生技已獲其許可進行重製與轉載分享。 References
|
|||

|
 |
||
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]
 具有堪比 NGS 準確度的 HiFi 長讀取定序技術,已廣泛地運用在
具有堪比 NGS 準確度的 HiFi 長讀取定序技術,已廣泛地運用在