|
看第三代定序技術「Iso-Seq - RNA 全長定序」如何協助發現新的癌症與疾病治療標靶及生物標記 |
|
|
自 1978 年 Walter Gilbert 首先提出單一基因可能經由選擇性剪接 (alternative splicing) 產生多種 mRNA 序列的想法 [1],次世代 RNA 定序技術「RNA-seq」的出現證實了 90% 以上的人類基因會透過選擇性剪接機制產生多種轉錄異構物 (transcript isoforms) [2-4]。此發現,使得轉錄體 (transcriptome) 研究變為益加重要與複雜。 然而隨著 isoforms 研究需求的增加與深入,RNA-seq 技術逐漸無法滿足科研人員。《Nature Methods》期刊曾將轉錄體比喻為一家書店,使用 RNA-seq 定序轉錄體,如同將書店中所有雜誌(RNAs)送入碎紙機中,再利用膠帶(演算軟體)嘗試將這些碎紙(序列片段)拼湊還原為一本本的雜誌(圖 1)[5]。在這拼湊過程中,長度僅有短短 100 ~ 300 nt 的 RNA-seq 定序數據 (read) 通常僅能涵蓋兩個鄰近外顯子 (exon) 的序列,高度仰賴演算軟體推斷組裝出的 isoforms 序列,往往極易產生錯誤造成結果的誤判 [2, 4, 6, 7]。
圖 1﹑《Nature Methods》期刊曾將轉錄體比喻為一家書店,使用 RNA-seq 定序轉錄體,如同將書店中所有雜誌(RNAs)送入碎紙機中,再利用膠帶(演算軟體)嘗試將這些碎紙(序列片段)拼湊還原為一本本的雜誌。 IMAGE © Nat Methods. 2013 Dec;10(12):1165-6. Figure 1 [5]. |
|
第三代定序技術「Iso-Seq - RNA 全長定序」幾乎不需組裝,即可完整呈現 isoform 全長序列 |
|
|
由 Pacific Biosciences (PacBio) 公司所開發的 RNA 全長定序技術「Iso-Seq (isoform sequencing)」兼具有 PacBio 獨家 HiFi 定序技術的高準確度 (>99.9%) 與第三代定序技術的長讀取 (long-read) 特性,單一定序數據 (single read) 就能完整涵蓋 10 kb 左右轉錄產物 (transcript) 的全長序列(從 5' 端至 3' poly(A) 尾端)。這使得大部分的 transcript isoforms 都能不經過組裝,自 Iso-Seq 測序結果中直接讀取到全長序列,大幅減少演算軟體的影響與干擾,精確地呈現各個 isoforms 的外顯子組合樣貌(圖 2)[8, 9]。
圖 2﹑Iso-Seq 定序技術兼具有高準確度與長讀取特性,幾乎不需組裝就能在單一定序數據中完整涵蓋轉錄產物的全長序列,精確地呈現各個 isoforms 的外顯子組合樣貌。 IMAGE © PacBio 2020 [8]. Iso-Seq 已成功地運用在多種物種(例如人類 [10, 11]、兔子 [12]、水稻 [13]、玉米 [14])以及組織細胞(例如大腦皮質 [15]、全血 [16]、胚胎幹細胞 [17]、眼角膜上皮細胞 [18])的轉錄體定序與基因體註解 (genome annotation) [19, 20]。
圖 3﹑佛羅里達大學 Ana Conesa 教授與愛丁堡大學羅斯林研究所 (Roslin Institute) Richard Kuo 先生分享他們使用 Iso-Seq 定序技術進行 isoforms 與長鏈非編碼 RNA (long non-coding RNA) 研究的經驗與感想,歡迎點擊觀看影片(片長約 1 分鐘)。 以下我們將透過幾個實際案例簡單介紹 Iso-Seq 如何參與癌症腫瘤與疾病醫療研究,協助發現新的治療標靶及生物標記。 |
|
Iso-Seq 協助發現新的乳癌治療標靶與病患存活率相關選擇性剪接事件 |
|
|
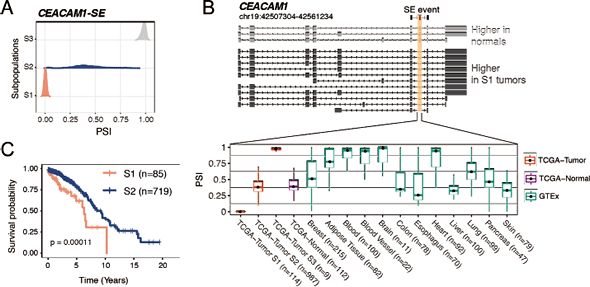
美國傑克遜基因組醫學實驗室 (The Jackson Laboratory for Genomic Medicine) Diogo F.T. Veiga 等人使用 Iso-Seq 定序技術分析 26 個人類乳癌樣本與 4 個正常乳房樣本的轉錄體,並將定序分析結果與 GENCODE v.30 資料庫進行比對,總共發現了 95,398 個新的 isoforms,其中 80% 都可以在 ATAC-seq、CAGE 或 3'-Seq 數據中獲得證實。 透過分析 isoforms 的轉譯序列,發現有許多乳癌相關基因的 isoforms 會發生蛋白質功能區域缺失或分布位置變化。例如 ESR1 (ERα) 基因就有 5 個新發現的 isoforms 其轉譯出的蛋白質序列缺乏 DNA 結合區域、11 個新發現的 isoforms 缺乏轉錄活性區域 (transactivation domain)、以及 1 個新發現的 isoform 具有穿膜區域,顯示該 isoform 的蛋白質座落位置會由原本的細胞核轉變為細胞膜,也意味著該 isoform 具備有成為癌症免疫療法標靶的可能性。 作者進一步藉由比對 1,135 個人類乳癌組織與 1,443 個正常組織選擇性剪接發生機率的差異性,找尋出 35 個與乳癌病患存活率相關的選擇性剪接事件。例如:依據 CEACAM1 isoforms 存留有 exon 7 的可能性將乳癌病患區分為 S1 ~ S3 三個亞群,其中 exon 7 存留率最低的 S1 亞群其病患存活率遠低於 exon 7 存留率較高的 S2 亞群,顯示 CEACAM1 exon 7 與乳癌病患存活率具有相關性(圖 4)[21]。
圖 4﹑(A) 依據 CEACAM1 isoforms 存留有 exon 7 的可能性將乳癌病患區分為 S1 ~ S3 三個亞群。(B) 無論是乳癌病患組織或一般正常組織皆有可能表現存留有 exon 7 的 CEACAM1 isoforms,然而可以明顯地觀察到 S1 病患亞群的 exon 7 存留率最低。(C) S1 亞群的存活率遠低於 S2 亞群,顯示 CEACAM1 exon 7 與乳癌病患存活率具有相關性。 IMAGE © bioRxiv 2020.11.10.376996. Fig. 5A-5C [21]. Diogo F.T. Veiga 等人的研究成果不僅有助於發現新的乳癌癌症免疫療法標靶,同時也為乳癌致病機轉與治療藥物抗藥性研究帶來許多新的線索與啟發。 |
|
Iso-Seq 精確檢測兒童 B 細胞急性淋巴性白血病相關 IGH-DUX4 融合基因全長序列,協助發現新的癌症治療標靶 |
|
|
根據統計,約有 7% 的兒童 B 細胞急性淋巴性白血病 (B-cell acute lymphoblastic leukemia, B-ALL) 病患表現有 IGH-DUX4 融合基因(以下簡稱 IGH-DUX4 fusion),該融合基因是由 DUX4 基因易位 (translocation) 至 IGH (immunoglobulin heavy chain, Igμ) 基因區域所形成,而 DUX4 基因具有轉錄因子功能,正常情況下僅會在人類胚胎卵裂 (cleavage) 時期表現;然而具有 IGH-DUX4 fusion 的兒童 B-ALL 病患,卻會不正常地表現 DUX4 基因。 美國聖裘德兒童研究醫院 (St. Jude Children's Research Hospital) Liqing Tian 等人發現到一個奇怪的現象,雖然病患的 DUX4 表現量遠高於一般正常卵裂時期胚胎,然而若是比較病患本身的 Igμ 與 DUX4 表現量,會發現 Igμ 表現量遠高於 DUX4。由於在 B 細胞發育過程中,IGH (Igμ) 基因存在有對偶基因剔除 (allelic exclusion) 現象,亦即兩條染色體中僅會有一個染色體的 IGH allele 活躍表達出 Igμ 蛋白,另一個 aelle 則受到調控靜默不表達,因此作者懷疑病患樣本中 Igμ 與 DUX4 表現量失衡的原因,是由於 DUX4 基因易位發生在靜默的 IGH allele 上所導致。 透過 Iso-Seq 定序技術,作者得以從帶有 IGH-DUX4 fusion 的 B-ALL 疾病模式細胞株「Nalm6」的轉錄體定序結果中取得 Igμ 以及 2 種 IGH-DUX4 fusion、1 個反義 (antisense) DUX4 產物的 RNA 全長序列。將這些 RNA 全長序列與作者組裝出的單倍型 (haplotype) 基因體序列進行比對,可輕易地區分出 Igμ 與 IGH-DUX4 fusion、antisense DUX4 是分別位在兩條不同的染色體上(圖 5)。基因體序列甲基化程度分析結果,也佐證了 IGH-DUX4 fusion、antisense DUX4 位處於靜默的 IGH allele。 透過 Iso-Seq 定序技術,作者得以順利地鑑別出 IGH-DUX4 fusion 座落於靜默的 IGH allele 上,也由此引導作者進一步假設在 IGH-DUX4 fusion B-ALL 族群中,或許 DUX4 的表現量必須經過精準調控,才能有助於癌化發生。經由大量表現 DUX4 的 Nalm6 細胞,也證實過量的 DUX4 會促使細胞凋亡 (apoptosis) ,顯示 DUX4 具有發展為 IGH-DUX4 fusion B-ALL 治療標靶的潛力 [22]。
圖 5﹑透過將 Iso-Seq 定序技術所得到的 RNA 全長序列與單倍型基因體序列進行比對(兩個單倍型序列分別帶有 T-G-G 與 A-A-A 的單一核苷酸變異 (SNPs) 特徵),可確認 IGH-DUX4 fusion、antisense DUX4 與 Igμ 是分別位在兩條不同的染色體上。 IMAGE © Nat Commun. 2019 Jun 26;10(1):2789. Fig. 2a [22]. |
|
Iso-Seq 發現新的遺傳性視網膜退化疾病相關 isoforms,協助建立正確的疾病模式動物 |
|
|
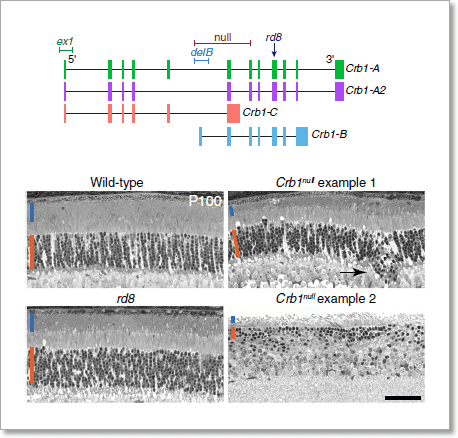
過去研究已證實,CRB1 基因突變與多種人類遺傳性視網膜退化疾病 (inherited retinal degenerations, IRD) 相關,例如萊伯氏先天性黑蒙症 (Leber congenital amaurosis, LCA)、視網膜色素變性 (retinitis pigmentosa, RP)、錐-桿狀細胞失養症 (cone-rod dystrophy, CRD) 等等。然而現有的 Crb1 基因突變小鼠卻無法很好地表現出應有的病理特徵,其感光細胞 (photoreceptors) 退化程度極為緩慢或趨近於無。 杜克大學醫學院 (Duke University School of Medicine) Thomas A. Ray 等人使用 Iso-Seq 定序技術從發育成熟的小鼠視網膜樣本中總共發現了 4 種 Crb1 isoforms,令人驚訝的是在視網膜中表現量最多的 Crb1 isoform,並非過去廣為研究的 Crb1 基因(為了方便區別,作者將此已知 isoform 命名為 Crb1-A),而是另一個從未曾被報導過的 Crb1-B isoform。在人類視網膜的 Iso-Seq 定序結果中,同樣可觀察到表現量最多的 CRB1 isoform 成員是新發現的 CRB1-B isoform 而非原本所認知的 CRB1 基因(亦即 CRB1-A isoform)。至此,作者合理地懷疑過去 Crb1 基因突變小鼠之所以效果不彰的原因,是因為忽略了 Crb1-B isoform 的存在。 從圖 6 圖示中可以看到,過去研究中所建立的 Crb1 基因突變小鼠(rd8)其點突變位置無法影響到所有 Crb1 isoforms,另一個曾報導過的 Crb1 基因剔除小鼠(ex1)所造成的基因改變更是完全無法影響到 Crb1-B isoform。作者使用 CRISPR/Cas9 技術建立了真正的 Crb1 基因剔除小鼠(null),可徹底破壞所有 Crb1 isoforms 的表現。視網膜組織電顯圖證實作者所建立的 Crb1null 小鼠,外核層 (outer nuclear layer, ONL)(圖 6 橘線標示處)厚度變薄、核數量也變得較為稀少,同時感光細胞外節 (outer segment)(圖 6 藍線標示處)的長度也明顯變短,顯示 Crb1null 小鼠更加適合作為研究人類遺傳性視網膜退化疾病的模式動物 [23]。
圖 6﹑視網膜組織電顯圖證實作者所建立的 Crb1null 小鼠,外核層(橘線標示處,為感光細胞的細胞核所在位置)厚度變薄、核數量也變得較為稀少,同時感光細胞外節(藍線標示處,為感光細胞的感光部分)的長度也明顯變短。 IMAGE © Nat Commun. 2020 Jul 3;11(1):3328. Fig. 9a and 10b [23]. Thomas A. Ray 等人的研究顯示 Iso-Seq 定序技術具有極佳的 isoforms 發現能力,同時也表明完整的 isoforms 資訊將有助於建立更完善的疾病模式動物。 |
|
使用 Iso-Seq 進行 Allele Phasing,協助鑑定重要的乳癌用藥指標 |
|
|
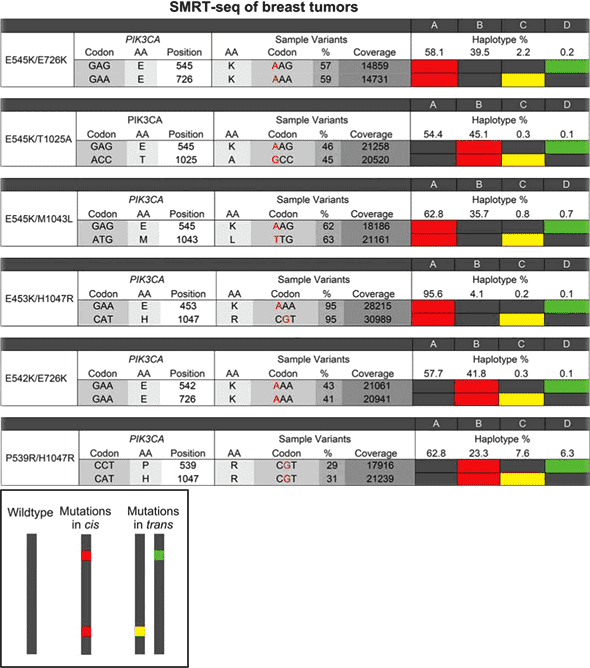
在人類癌症中 PIK3CA 基因是最常見的突變致癌基因。美國斯隆·凱特琳紀念癌症中心 (Memorial Sloan Kettering Cancer Center) Neil Vasan 等人發現有一群乳癌患者對於 PI3Kα 抑制藥物「alpelisib」具有較好的治療反應,這些病患體內的 PIK3CA 基因都帶有兩個序列位點突變(以下簡稱 double-PIK3CA-mutant),這個發現啟發作者進行更大範圍的研究調查。 在 70,754 個受測病患中(包含乳癌、大腸直腸癌、子宮內膜癌等各類型癌症病患),13% 的病患其 PIK3CA 基因帶有多個序列位點突變(以下簡稱 multiple PIK3CA mutations)。聚焦分析乳癌這個族群,約有 12-15% 的乳癌病患具有 multiple PIK3CA mutations,其中高達 95% 的案例是屬於 double-PIK3CA-mutant。而以乳癌亞型來看,multiple PIK3CA mutations 主要是發生在雌激素受體陽性且人類表皮生長因子受體陰性 (ER+/HER2-) 這個族群裡。 為了鑑別乳癌病患所帶有的 double-PIK3CA-mutant,是發生在同壹條 PIK3CA allele 的順式突變 (mutations in cis),還是兩條 PIK3CA alleles 各帶有一個序列突變的反式突變 (mutations in trans),作者使用 Iso-Seq 定序技術進行對偶基因定相 (allele phasing) 分析。從 6 個帶有 double-PIK3CA-mutant 的乳癌患者 PIK3CA RNA 定序結果來看,所有 double-PIK3CA-mutant 皆是以順式突變的方式存在(圖 7),這個資訊對於建立精確的臨床醫學生物標記極為重要。 作者也透過 508 位 ER+ 轉移性乳癌病患的用藥實驗證實,帶有 multiple PIK3CA mutations 的病患,相較於僅帶有單一 PIK3CA mutation 或是不具有 PIK3CA mutation 的病患,對於 PI3Kα/γ/δ 抑制藥物「taselisib (GDC-0032)」的治療反應更佳,顯示 PIK3CA mutations 資訊可作為乳癌患者臨床用藥的重要參考指標 [24]。
圖 7﹑使用 Iso-Seq 定序技術分析 6 個乳癌病患 PIK3CA 基因的 RNA 序列,結果顯示病患所帶有的 double-PIK3CA-mutant 座落在同壹條對偶基因上,為順式突變。 IMAGE © Science. 2019 Nov 8;366(6466):714-723. Fig. 1F [24]. 若您想更進一步深入了解 Iso-Seq,歡迎報名參加即將在 3 月 3 日登場的 Iso-Seq 中文線上講座〈Using the Iso-Seq Method For Bulk and Single-Cell Isoform Sequencing〉,由 PacBio 公司首席研究員 Elizabeth Tseng 博士主講,誠摯邀請您共同參與交流討論 RNA 定序的相關應用進展與實踐問題。更多 Iso-Seq 應用實例與完整產品資訊,歡迎洽詢 PacBio 台灣代理 — 伯森生技。 References
|
|

|
 |
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]