
|
《Genomics》期刊論文選讀 以 HiFi 定序技術破解全外顯子定序未能檢測出的併發症型智能障礙基因變異 以次世代定序技術 (next-generation sequencing, NGS) 為基礎的全外顯子定序 (whole-exome sequencing, WES) 與全基因體定序 (whole-genome sequencing, WGS) 在近十年左右被大量地使用在遺傳疾病與基因體變異的臨床分子診斷,然而平均不到五成的診斷率意味著仍有許多病患亟需其他診斷方法的介入來幫助找出確切病因以擬定有效的治療方案 [1, 2]。 日本橫濱市立大學醫學研究所 Naomichi Matsumoto 教授研究團隊在《Genomics》期刊就詳盡報導了一對同卵雙胞胎女童病例,這對雙胞胎從臨床症狀上判斷疑似罹患了卓飛症候群 (Dravet syndrome),然而使用全外顯子定序分析其中一個女童病患與其父母三人的基因體序列卻無法找到任何可能的致病變異,於是 Naomichi Matsumoto 團隊轉向尋求第三代定序技術的幫助 [3]。
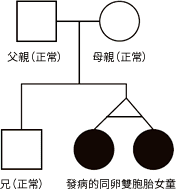
圖 1﹑病患家庭成員組成與臨床表徵簡述。發病的同卵雙胞胎女童是由非近親關係的父母所生育,女童們的父母與兄長皆正常無任何病徵。在病患們出生五個月後發現有動作發展遲緩徵兆,在一歲左右可觀察到眼瞼下垂、眼距過寬、眼內貲贅皮、斜視、寬鼻樑、人中短小等明顯的面部畸形特徵。一歲半後開始出現癲癇,並在五、六歲時透過電腦斷層掃描 (CT) 發現部分頸椎有融合現象。12 歲時表現有語言發展遲緩與中度智能障礙,腦電圖 (EEG) 呈現多處局部棘波 (multi-focal spikes) 與棘慢波放電 (spike-and-wave discharges),磁振造影 (MRI) 檢查正常、一般血液與生化檢查顯示無代謝異常。兩個女童的臨床表徵極為相似,顯示兩者極有可能帶有同樣的基因體變異。 IMAGE © Genomics. 2020 Nov 4:S0888-7543(20)31997-2. Fig. 1a [3]. |
|
使用 HiFi 定序技術成功鑑定找出 12 kb 大小的致病性倒轉基因變異 |
|
|
Naomichi Matsumoto 團隊所選擇使用的第三代定序技術是 Pacific Biosciences (PacBio) 公司所開發的「HiFi 定序技術」。HiFi 定序兼具有第三代定序技術的長讀取 (long-read) 特性(平均定序長度為 13.5 kb)與媲美 NGS 的高準確度 (high fidelity)(平均準確度為 99.8%)[4]。對於人類基因體單核苷酸變異 (SNVs, 1 bp)、插入缺失變異 (Indels, <49 bp) 與結構變異 (SVs, ≥50 bp),HiFi 定序都具有非常出色的檢測能力。例如在最新一屆的全球 precisionFDA 真實挑戰賽中,HiFi 定序技術與 DeepVariant 分析軟體的組合就從眾多競爭者中脫穎而出,成為變異檢測準確度最佳的定序技術與分析策略組合,其檢測錯誤數量比起當前主流的 Illumina 與 GATK 組合少了 5.8 倍、比 Illumina 與 DeepVariant 組合少了 2.5 倍 [5];此外,華盛頓大學醫學院 Mark J.P. Chaisson 等人所進行的定序技術評比實驗,也證實了 PacBio 長讀取定序技術對於人類基因體結構變異的檢測能力遠勝過 NGS 短讀取定序技術,特別是在重複序列區域 [6]。
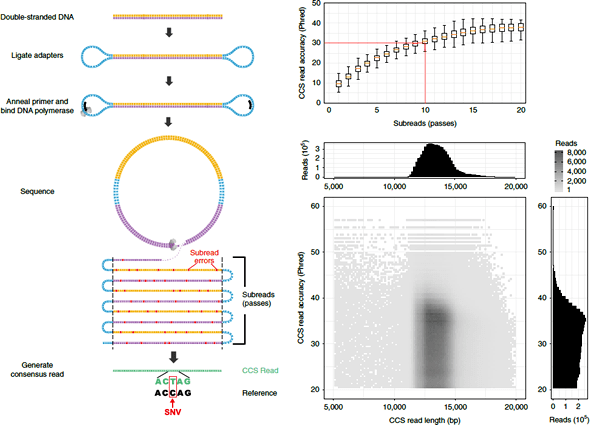
圖 2﹑HiFi 定序原理。在進行 HiFi 定序時,待測 DNA 序列兩端會先銜接上一段序列 (adapter) 使其形成環狀 DNA。透過 PacBio 定序系統獨有的循環共識定序模式 (circular consensus sequencing, CCS),待測 DNA 將會被反覆測序。由於 PacBio 定序技術不具有系統性錯誤,因此可以藉由比對反覆測序的序列結果將隨機產生的定序錯誤加以修正,並保留下真正的基因體變異數據,最終達到 Q30 的高準確度。HiFi 定序的平均定序長度為 13.5 kb。 IMAGE © Nat Biotechnol. 2019 Oct;37(10):1155-1162. Fig. 1 [4]. Naomichi Matsumoto 團隊使用 HiFi 定序技術搭配 Sequel II 定序系統對其中一個女童病患與其父母三人進行全基因體定序,接著將定序結果與 hg19 人類參考基因體進行序列比對,並使用 pbsv 軟體檢測結構變異、以 DeepVariant 軟體檢測單核苷酸變異與插入缺失變異,最終在女童的第三號染色體找到一段長 12 kb 的倒轉 (inversion) 結構變異。這段倒轉序列是由 BRPF1 基因的前 2 個外顯子 (exon) 與 CPNE9 基因的最後四個外顯子所組成。由於倒轉變異將會導致這兩個基因失去功能,因此作者進一步查詢比對 GnomAD 資料庫,發現 BRPF1 基因的不耐受喪失功能突變指數 (probability of being loss-of-function intolerant, pLI) 為 1、CPNE9 基因則為 0,顯示 BRPF1 基因的功能喪失突變極有可能就是女童罹病的主要病因。更重要的是,女童臨床症狀與 OMIM 資料庫中所記載的 BRPF1 基因變異相關疾病“智能障礙伴隨面部畸形與眼瞼下垂 (intellectual developmental disorder with dysmorphic facies and ptosis, IDDDFP, OMIN #617333)”的臨床特徵高度相符,顯示女童所罹患的疾病是由此 12 kb 倒轉基因變異所導致的併發症型智能障礙 (syndromic intellectual disability, syndromic ID),而非原本所推測的卓飛症候群。
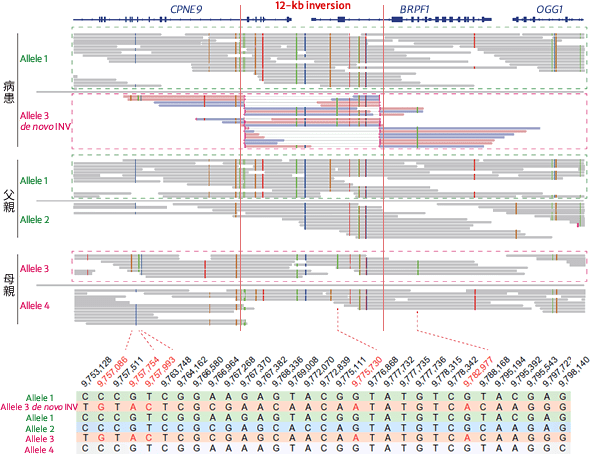
圖 3﹑使用 HiFi 定序結果進行單倍體定相 (haplotype phasing) 分析。由於 HiFi 定序結果具有高準確度,因此可以藉由序列中的單核苷酸變異與插入缺失變異特徵進行單倍體定相。分析結果顯示,女童所帶有的 12 kb 倒轉基因變異是發生在源自於母親的那壹條第三號染色體上,且為患者自己本身的新生突變 (de novo mutation)。 IMAGE © Genomics. 2020 Nov 4:S0888-7543(20)31997-2. Fig. 3b [3]. Naomichi Matsumoto 團隊表示,此次所發現的12 kb 致病性倒轉基因變異由於沒有造成拷貝數變化 (copy neutral),且變異發生位置是座落在重複序列之間,這些因素都是導致團隊之前使用外顯子定序無法檢測得到的原因。除此之外,目前的染色體檢測技術顯然解析度不足(約 10 Mb 左右),也無法檢測出這類僅有數 kb 大小的結構變異;使用 HiFi 定序技術將能夠彌補現行檢測技術的不足,提升疾病的診斷率。
References
|
|

|
 |
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]
 近期美國堪薩斯城兒童慈善醫院就與 PacBio 和 Microsoft 共同合作展開「
近期美國堪薩斯城兒童慈善醫院就與 PacBio 和 Microsoft 共同合作展開「