|
從《Nature Genetics》論文看 HiFi 定序技術如何參與馬鈴薯育種計畫 |
|
|
馬鈴薯 (Solanum tuberosum L.) 是全球最重要的塊莖類作物。與其他主要農作物不同,馬鈴薯的栽種主要仰賴種薯的無性繁殖,這種種植方式不僅需要花費大量的時間空間運送保存種薯,且由於栽培產物屬於同源四倍體的關係,使得遺傳訊息複雜、品種改良不易。 為縮短馬鈴薯品種改良週期與提高作物經濟效益,中國農業科學院深圳農業基因組研究所黃三文教授研究團隊計畫以二倍體雜交種子的有性繁殖方式取代傳統四倍體種薯無性繁殖的栽培方式。2018 年,黃三文團隊首先利用 CRISPR-Cas9 技術使 S-RNase 基因突變喪失功能,攻克了二倍體馬鈴薯普遍存在的自交不親和性 (self-incompatible) 問題 [1];接著在 2019 年,黃三文團隊進一步找尋出二倍體馬鈴薯近交衰退 (inbreeding depression) 的相關基因區域,並從潛在的隱性有害基因中鑑定找出五個致命突變基因與四個影響作物長勢的基因,此發現對於後續雜交種子的基因圖譜設計具有重大意義 [2]。
圖 1﹑二倍體馬鈴薯 RH (RH89-039-16)。 緊接著在 2020 年 9 月 28 日,黃三文團隊在《Nature Genetics》雜誌線上發表了異型合子 (heterozygous) 二倍體馬鈴薯(品系 RH89-039-16,以下簡稱 RH)的全基因體定序與分析結果,為後續二倍體馬鈴薯的基因改造工作奠定了良好的基石。該研究成果不僅是目前最為完整的二倍體馬鈴薯基因體資訊,也是第一個能夠完全解析二倍體馬鈴薯兩套單倍體基因型 (haplotype) 的案例 [4]。以下就讓我們來看看黃三文團隊如何進行 RH 馬鈴薯的從頭定序 (de novo sequencing) 與單倍體定相 (haplotype phasing)。
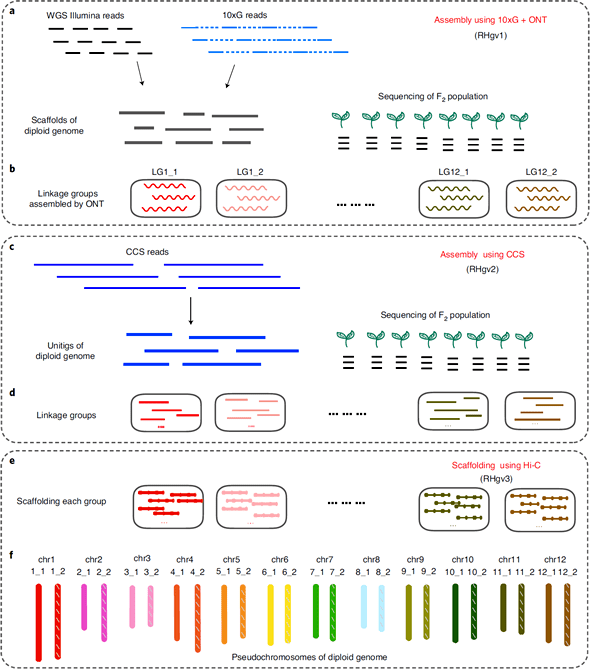
圖 2﹑黃三文團隊於《Nature Genetics》雜誌發表了全球首例的 RH 馬鈴薯完整基因體序列與單倍體基因型定相資訊。IMAGE © Nature Genetics [4]. 起初,黃三文團隊試著使用 Oxford Nanopore Technologies (ONT) 公司的長讀取 (long read) 定序技術結合 Hi-C (high-throughput/resolution chromosome conformation capture) 染色體結構捕捉技術的定序結果來進行 RH 基因體的序列組裝與定相,然而結果卻不甚理想。最終,則是藉由三個階段的定序組裝與序列分析策略,整合完成 12 對染色體、24 個單倍體的定序與定相工作。 在第一個階段,使用了 Illumina 與 10x Genomics (10xG) 的全基因體定序數據進行序列組裝,再藉由 RH 馬鈴薯自交子代 (F2) 的定序結果協助進行單倍體定相,如此可分離出 24 條、共 12 對染色體的序列,然後再利用 ONT 與 10xG 的定序結果進行精細校正,至此可得到第一階段的全基因體定序結果,稱之為 RHgv1,序列總長 1.69 Gb,組裝出的序列骨架 (scaffold) N50 長度為 920 kb(圖 3a, 3b)。 在第二個階段,全基因體定序數據改採用以 PacBio 公司循環共識定序模式 (Circular Consensus Sequencing, CCS) 所產生出的 HiFi 定序數據進行序列組裝,同樣藉由 RH 馬鈴薯自交子代 (F2) 的定序結果協助進行單倍體定相,亦可分離出 24 條、共 12 對染色體的序列,此為第二階段的全基因體定序結果,稱之為 RHgv2,序列總長 1.31 Gb,組裝出的序列骨架 N50 長度為 2.19 Mb(圖 3c, 3d)。 作者還進一步使用先前所完成的 BAC 定序結果來驗證評估 RHgv1 與 RHgv2 的品質。在 184 個組裝出的 BAC 序列片段中,分別有 126 與 169 個 BAC 序列片段可以匹配至 RHgv1 與 RHgv2 的單一片段上;從 BAC 匹配序列準確度來看,RHgv1 與 RHgv2 的序列準確度分別為 99.127% 與 99.936%。整體而言,無論是在序列的連續性或準確度,RHgv2 都比 RHgv1 要更加優越。 最後作者整合了 RHgv1 與 RHgv2,協同 Hi-C 定序結果,產生出最終第三階段的全基因體(共 12 對染色體、24 個單倍體)的序列資訊,稱之為 RHgv3,序列總長 1.62 Gb(圖 3e, 3f)。
圖 3﹑黃三文團隊整合不同定序技術數據,組合定相出 RH 馬鈴薯全基因體(共 12 對染色體、24 個單倍體)的序列資訊,總長 1.62 Gb。 IMAGE © Nat Genet. 2020 Oct;52(10):1018-1023. Fig. 1 [4]. 作者在文中特別提到了高準確度的 HiFi 長讀取定序數據(即文中所指的 CCS reads)對於解析複雜基因體的能力優於短讀取定序技術以及較易出錯的 ONT 長讀取定序技術,作者寫道 "Compared with short reads or the longer but more error-prone ONT reads, CCS reads generated higher resolution and accuracy in differentiating haplotypes, which is particularly useful in resolving complex genomes." [4]。 無獨有偶,美國鮑依斯湯普森研究所 (Boyce Thompson Institute) 費章君教授研究團隊以及美國農業部國家農業研究局 (USDA-ARS) 仲幹遠教授研究團隊在其所合作的研究專案中同樣選擇使用 PacBio HiFi 定序技術來協助完成栽培種蘋果 (Malus domestica cv. Gala) 與其兩種野生祖先種蘋果(M. sieversii 與 M. sylvestris)的從頭定序與單倍體定相工作;並進一步將 HiFi 定序技術應用於這三類蘋果的泛基因體 (pan-genome) 定序,以了解蘋果馴化過程中基因體變化與性狀改變的關連性。其研究成果已線上發表於《Nature Genetics》雜誌網站 [5]。 無論是黃三文教授或是費章君教授等人的研究,都在在顯示 PacBio HiFi 定序技術在現代農業作物育種領域的卓越效能與潛力。您可以透過這個網頁瀏覽更多 HiFi 定序技術於動植物學術領域的實際應用案例。完整產品資訊與最新活動訊息,歡迎洽詢 PacBio 台灣代理 — 伯森生技。 References
|
|

|
 |
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]