|
🚩 別錯過人類基因體序列的重要里程碑 看 T2T 聯盟如何完成史上第一個“完整的”人類基因體序列
有些人或許不知道,由美國能源部 (DOE) 和國家衛生研究院 (NIH) 在 1990 年共同主導展開的跨國性「人類基因體計劃 (Human Genome Project, HGP)」雖然已在 2003 年宣布完成,然而其實我們一直以來都沒有真正獲得過“完整的”人類基因體序列。以目前大家使用的人類基因體參考序列 GRCh38.p13 為例,它是在 2013 年由參考基因體協會 (Genome Reference Consortium, GRC) 釋出,最後更新時間為 2019 年;GRCh38.p13 缺少了近 8% 的序列,這些缺失主要分布在異染色質 (heterochromatin) 與複雜區域,包含著絲粒衛星陣列 (centromeric satellite arrays)、rDNA 陣列、次端粒 (subtelomeric) 區域等,最明顯的缺口就落在近端著絲點染色體 (acrocentric chromosomes) ——也就是第 13, 14, 15, 21, 22 號染色體——這五個染色體的整個短臂 (p-arms) 序列在 GRCh38.p13 中通通都缺失了!可以發現這些缺失的部分幾乎都具有重複序列特性,無論是桑格定序 (Sanger sequencing) 或次世代定序 (NGS) 都無法有效觸及與覆蓋,因此長期以來都處於序列不明或未知的狀態。 端粒到端粒聯盟 (Telomere to Telomere Consortium, 以下簡稱 T2T 聯盟) 最新釋出的人類基因體序列 CHM13v1.1,不僅彌補了 GRCh38.p13 所有的序列缺失,也校正了許多原本的組裝錯誤,最終完成全長約 30.55 億個鹼基對 (bp) 的全基因體序列,也是史上第一個沒有任何間隙 (gap)、完整且連續的人類基因體序列!值得一提的是,CHM13v1.1 含有超過 1.8 億個在 GRCh38.p13 找不到的全新序列,裡面蘊含了 2,226 個旁系同源基因 (paralogous genes),估計有 115 個基因會轉譯出蛋白質。這些珍貴的全新發現基因將連同所有新揭露的困難與複雜區域完整序列資訊,為生物醫學研究帶來極佳助益。您可以透過 T2T 聯盟發表於《bioRxiv》的文章,更深入地了解 GRCh38.p13 與 CHM13v1.1 之間的差別。 以下,我們則將帶您進一步來看 T2T 聯盟是如何跨越困難區域的重重阻礙,完整組裝出人類基因體序列?
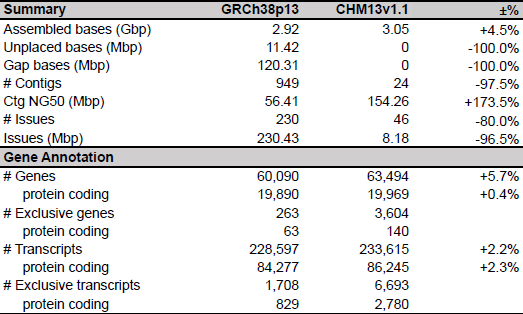
表 1﹑人類基因體參考序列 GRCh38.p13* 與 T2T 聯盟最新完成的人類基因體完整序列 CHM13v1.1 比較表。 *GRCh38p13 summary statistics exclude "alts" (110 Mbp), patches (63 Mbp), and Chromosome Y (58 Mbp). IMAGE © bioRxiv. 2021 May 27. DOI: 10.1101/2021.05.26.445798. Table 1.
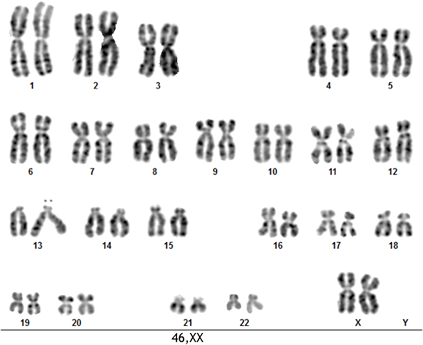
1. 選擇 CHM13hTERT 細胞株作為定序樣本,以降低組裝複雜度 CHM13hTERT 是一種源自於人類完全性葡萄胎 (complete hydatidiform mole, CHM) 的細胞株,染色體核型 (karyotype) 為 46,XX,亦即 22 對體染色體 (autosomes) 加上 1 對 X 染色體。由於它的染色體全是由精子本身的單套染色體複製而來,因此可視為是近乎完全一致的同型合子 (homozygous)。使用 CHM13hTERT 作為定序樣本,一來可以降低組裝複雜度,再來是可以確保組裝出的單倍體 (haplotype) 序列是源自於單一樣本(※人類基因體參考序列 GRCh38.p13 則是由數個捐贈者樣本的定序數據混雜組裝而成)。
圖 1﹑CHM13hTERT 細胞的染色體核型 (karyotype)。 2. 使用 PacBio HiFi 定序數據作為組裝基礎 由 PacBio 公司所開發的 HiFi 定序是 T2T 聯盟達成人類基因體完整組裝的重要關鍵之一!首先,不可諱言地,T2T 聯盟起初其實是採用奈米孔超長定序數據作為組裝基礎的,然而後來發現,雖然極長數據易於組裝,但是錯誤率太高,使得他們不得不花費大量時間與心力,以其他定序技術數據來進行錯誤糾正。這種以高錯誤率數據為開端的組裝策略,明顯在效率上無法讓 T2T 聯盟滿意。PacBio HiFi 定序的出現則徹底打破了科學家們對第三代定序技術高錯誤率的既定印象,它能夠在提供長讀取數據的同時,還兼具有比肩 NGS 的高精準度 (>99.9%)。這種前所未見的雙重優勢,讓 T2T 聯盟決定轉向使用 HiFi 定序數據作為組裝基礎,建構形成序列骨架 (scaffold)。
此外,T2T 聯盟聯合主席 Karen Miga 博士在接受《Nature》期刊採訪時,也特別提到 HiFi 定序對於 T2T 聯盟完成此項壯舉的重要性。
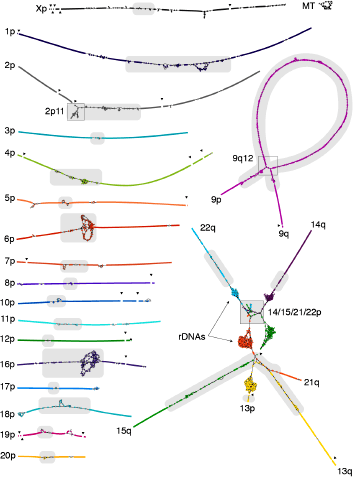
HiFi 定序能夠消弭技術藩籬,深入其他定序技術難以檢測的重複序列區域,使高度重複的著絲粒與端粒周邊區域、rDNA 陣列和片段重複 (segmental duplications) 等困難區域的序列得以被 T2T 聯盟完整揭曉。不僅如此,HiFi 定序數據的高準確度,讓 T2T 聯盟在單純使用 HiFi 定序數據的狀況下就能組裝出精準度極高的序列骨架(圖 2),大幅降低後續驗證除錯的複雜度。最終完成的人類基因體序列 CHM13v1.1 準確度高達 Q70 左右,這個結果遠遠超出了 T2T 聯盟當初所設立的 Q40 目標。
圖 2﹑以 HiFi 定序數據組裝出的人類基因體示意圖。T2T 聯盟直接使用 HiFi 定序數據進行組裝,可以看到在未經過任何其他定序技術數據驗證除錯的狀態下,就能清楚地分出每壹條染色體,且大部分皆為線性結構。其中五個近端著絲點染色體(染色體 13, 14, 15, 21, 22)由於彼此間的序列有高度重合相似之處(特別是第 14, 15, 21, 22 號染色體的短臂),使得它們集結為一個群組。 IMAGE © bioRxiv. 2021 May 27. DOI: 10.1101/2021.05.26.445798. Fig. 2A.
為了填補空缺的人類 Y 染色體序列資訊,T2T 聯盟正在進行 HG002 細胞株(染色體核型為 46,XY)的定序與組裝工作,相信再過不久所有人類染色體序列都將被我們所知道。然而這並不是我們探索人類基因體的終點。接下來人類泛參考基因體聯盟 (Human Pangenome Reference Consortium) 將會複製 T2T 聯盟的成功經驗,共同合作在接下來三年內完成超過 300 人的定序、組裝與單倍體定相 (haplotype phasing),以更深入了解不同個體與種族間的基因體多樣性,向精準醫學的目標更往前邁進一步。 PacBio 全新推出的 Sequel® IIe 定序系統能夠讓您直接取得高品質的 HiFi 定序數據,協助您以更高效經濟的方式完成精確的全基因體定序 (whole genome sequencing, WGS) 與從頭組裝 (de novo assembly)。若您希望進一步了解 PacBio HiFi 定序實驗實踐細節與生物資訊分析流程,歡迎洽詢 PacBio 台灣代理 — 伯森生技。 您可透過下方連結瀏覽更多相關資訊:
References
|
||||||
 |
 |
|||||
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]