|
從「地球生物基因體計畫」看見 PacBio HiFi 定序在植物-動物-昆蟲全基因體定序的強大效能
你知道嗎?從 1970-2016 不到 50 年間,全球脊椎動物數量大幅減少了 68% [1],目前有超過 37,000 種物種正面臨滅絕危機 [2],這些數據清楚地告訴我們,氣候變遷與人類活動已為地球生態帶來災難性的影響。 為保護生物多樣性、實現永續生態發展並增進人類福祉(包含探索新型藥物與生質能源等),美國加州大學戴維斯分校 Harris Lewin 教授與多位美國專家學者,聯合巴西聖保羅研究基金會 (FAPESP) Marie-Anne van Sluys 教授與中國華大基因 (BGI) 周欣教授,於 2015 年共同草擬出「地球生物基因體計畫 (Earth BioGenome Project, EBP)」,預計以十年時間完成地球所有真核生物 (eukaryotes) 的全基因體定序 (whole-genome sequencing, WGS),並將特別針對瀕危物種優先執行,以幫助儘早訂定相關保護法規,減緩甚至阻止物種滅絕 [3, 4]。 點擊圖片了解地球生物基因體計畫成立宗旨與目標。
2018 年地球生物基因體計畫開始實施之際,所面臨到的是在 150 萬種已知真核生物中僅有不到 0.2% 的物種基因體序列為我們所知,要在 10 年間完成所有真核生物物種的全基因體定序,如同生物界的「登月計畫」——立意高遠卻也極具挑戰 [3]!所幸目前已有 17 個國家、44 個產學機構共同合作,總計 47 項大型基因體計畫參與聯盟 [5, 6],其中 Genome 10K 計畫更肩負著最佳化組裝流程與制定品質標準的重要使命 [3, 7]。 根據 Genome 10K 團隊去年四月發表在《Nature》期刊的最新研究成果論文,可以發現 PacBio 長讀取定序技術 (PacBio long-read sequencing technology) 為 Genome 10K 團隊組裝出參考基因體 (reference genome) 品質的完整基因體序列帶來極佳助益,特別是在重複序列、高 GC 含量等困難區域的定序與單倍體定相 (haplotype phasing) 分析上,更是起著極為關鍵不可或缺的作用 [7]。 點擊圖片閱讀 Genome 10K 團隊於《Nature》期刊發表的最新研究成果論文。
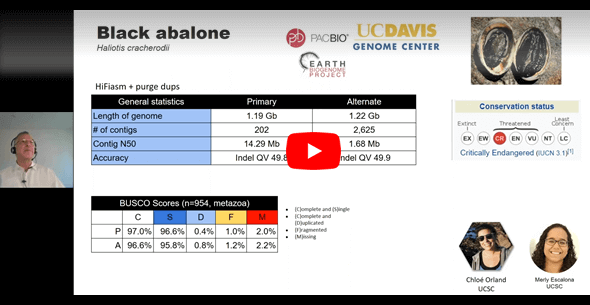
隨著 PacBio 長讀取定序技術——或稱為 SMRT 單一分子即時定序技術 (Single Molecule, Real-Time Sequencing)——試劑與儀器性能的提升,誕生出一種全新的具有高準確度的長讀取資料類型 “HiFi reads”。HiFi reads 不但能提供比肩 NGS 技術的高精準度 (>99.9%),還具有長讀取特性 (可達 25 kb),能完美補足 NGS 的短處。地球生物基因體計畫總主持人加州大學戴維斯分校 Harris Lewin 教授在去年一場線上演講中,即藉由「加州保育基因體計畫 (California Conservation Genomics Project, CCGP)」的近期成果向大家展示 PacBio HiFi 定序在全基因體定序的強大效能 [8]。 點擊圖片觀看地球生物基因體計畫總主持人 Harris Lewin 教授線上演講影片「New Discoveries in Plant and Animal Sciences」 *Harris Lewin 教授演講內容自 10:14 開始。 IMAGE © PacBio Webinar [8]. 例如,上圖中所顯示的是被國際自然保育聯盟 (IUCN) 列為極危 (Critically Endangered, CR) 物種的黑鮑螺 (Haliotis cracherodii) 的 HiFi 定序數據全基因體組裝結果。可以看到組裝出的兩套單倍體基因體序列皆具有極高的完整度,BUSCO 值分別為 97.0% 與 96.6%,Contig N50 長度分別是 14.29 Mb 與 1.68 Mb,準確度高達 QV49.8 以上,是非常高品質的全基因體組裝結果。其他在演講中分享的定序組裝成果還包含褐小灰蝶 (Brephidium exilis, 世界上最小的蝴蝶之一)、花蜆蝶 (Apodemia mormo virgulti, 瀕危物種)、百合 (Vellozia tubiflora) 與大樹鼩 (Tupaia tana) 等 [8]。
PacBio 長讀取定序技術不僅參與了前文所提的 Genome 10K 與加州保育基因體計畫,尚有多項地球生物基因體計畫的聯盟計畫也採用了 PacBio 定序技術,包含英國達爾文生命之樹 (Darwin Tree of Life)、美國百種農業害蟲 (Ag100 Pest (USDA))、澳洲瀕危物種倡議組織 (Threatened Species Initiative, TSI)、澳洲哺乳類基因體計畫 (Oz Mammals Genomics, OMG) 與跨國性脊椎動物基因體計畫 (Vertebrate Genomes Project, VGP) 等 [8, 9]。同時,從《Cell》、《Nature》、《Science》等多篇頂尖期刊文獻中 [10-20],我們也能窺見 PacBio 為植物、動物、昆蟲基因體研究與物種保存帶來的卓越助力,顯示 PacBio HiFi 定序技術已成為當代全基因體定序不可或缺的中堅力量!
PacBio 全新推出的 Sequel® IIe 定序系統能夠讓您直接取得高品質的 HiFi 定序數據,縮短 70% 的生物資訊分析時間,並減少 90% 的數據傳輸時間和儲存空間需求,協助您以更高效經濟的方式完成精確的全基因體定序與從頭組裝 (de novo assembly)。除此之外,PacBio 團隊還開發設計出三種不同 DNA 起始量需求的建庫流程,確保從多倍體生物到體型微小生物,都能得到最佳化的建庫指引。您可以透過這個影片快速了解如何選擇合適的建庫流程,同時也將看到數個應用實例的全基因體組裝數據,包含加州紅杉樹、蝴蝶、蚊子等等。若您希望進一步了解 PacBio HiFi 定序實驗實踐細節與生物資訊分析流程,歡迎洽詢 PacBio 台灣代理 — 伯森生技。 您可透過下方連結瀏覽更多相關資訊:
References
|
|||||
 |
 |
||||
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]